Os alvos wigplly conhecidos pelos cientistas como “proteínas intrinsecionadas” têm há décadas a captura por medicamentos e anticorpos personalizados. Mas eles desempenharam papéis biológicos tão importantes – ativando os receptores opióides; provocando mau comportamento da proteína associada à neurodegeneração; matar células produtoras de insulina – que os pesquisadores mantiveram atrás deles.
Agora, os cientistas de um laboratório da Universidade de Washington, liderados pelo ganhador do Nobel, David Baker, quebraram o desafio, usando IA generativa para criar proteínas que agarram as moléculas de mudança de forma. A descoberta pode desbloquear um conjunto de novos medicamentos e ferramentas de diagnóstico.
Quase metade das proteínas encontradas nos seres humanos é intrinsecamente desordenada: “No entanto, não tivemos maneira confiável de droga [them]”Baker disse em comunicado.“ Esses estudos mudam que, ao dar aos cientistas em todos os lugares novas ferramentas para vincular a metade não estruturada da biologia. ”

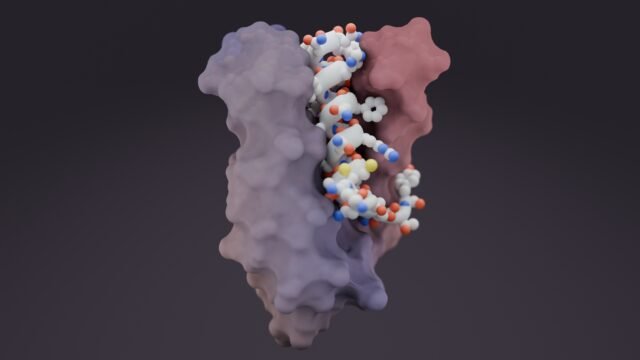
Ao contrário das proteínas típicas que se dobram em formas definidas, as proteínas intrinsecamente desordenadas são mais parecidas com espaguete cozido – elas são flexíveis e não têm uma estrutura estável.
Os cientistas da UW construíram uma biblioteca de partes de proteínas que podem ser costuradas e aplicadas a diversos alvos, concentrando -se em curtos trechos de aminoácidos para a ligação.
Os pesquisadores testaram suas proteínas feitas sob medida com resultados promissores: um sinais de dor bloqueados com sucesso nas células humanas cultivadas, enquanto outro grupo de proteínas dissolvido ligado ao diabetes tipo 2. A técnica também se mostrou útil para detectar e marcar moléculas escassas, mas importantes, incluindo um marcador de doença examinado nos recém -nascidos.
Os resultados foram impressionantes – as proteínas do designer prenderam com sucesso em 39 dos 43 alvos testados, uma taxa de sucesso de 91%. Cada proteína se dobra com precisão em torno do alvo pretendido, criando um abraço apertado e específico.
A nova abordagem das proteínas de engenharia é descrita em dois artigos, um publicado hoje no diário Ciênciae um segundo disponível como um pré -impressão. A maioria de mais de duas dúzias de autores é do Departamento de Bioquímica da UW e do Instituto de Projeto de Proteínas da Universidade, liderado por Baker.
Pesquisadores em todo o mundo podem acessar o software program de código aberto on-line.

Kejia Wuum ex-aluno de pós-graduação do Baker Lab e agora bolsista de pós-doutorado, foi um co-líder do The Recosly Publicado Ciência papel ao lado Hanlun Jiang e Derrick Hicks. O projeto foi emocionante, disse Wu, porque period muito difícil e forneceu “espaço para ter pensamentos criativos”.
A maioria dessas idéias é um absurdo e falhará, disse Wu. “Mas então você será capaz de restringir [it] Para baixo – um de seus pensamentos pode fazer sentido, e você começa a trabalhar nisso “, continuou ela.” Então essa é a parte que eu mais gosto. Você pode criar métodos não tradicionais, pensamento não tradicional. ”
E, embora tenha sido difícil inicialmente criar estratégias de uma solução para as proteínas intrinsecionadas, dada sua natureza sem forma, essa fluidez tem uma vantagem.
Uma proteína dobrada e estruturada normalmente possui apenas uma solução para uma proteína de ligação, disse Wu. “Mas a plasticidade conformacional … na verdade nos dá liberdade para segmentar [the molecule] Muitas maneiras diferentes – e precisamos apenas de um deles para trabalhar. ”